NGS(DNA-Seq)
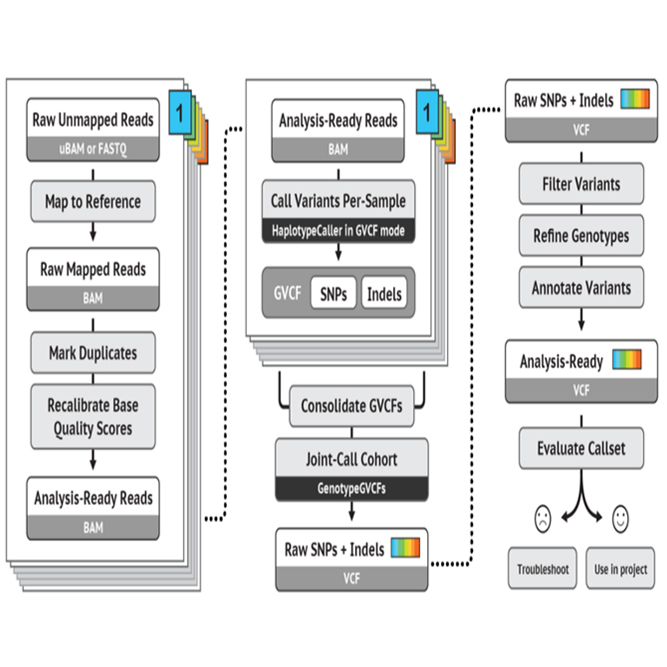
Genome Sequencing이 완료된 생물종을 대상으로 gDNA를 분리한 후 NGS 기술을 이용하여 전장유전체 분석을 수행해 드립니다. 기본 분석으로써 SNP, INDEL, CNV, Structural variation 분석 데이터를 제공하고 이외 다양한 분석을 지원해 드립니다.
Service Info.
| Sample requirement | >2ug gDNA |
|---|---|
| Library method | Illumina TruSeq Nano DNA Library kit |
| NGS run format | NovaSeq 6000, PE150 |
| Data yield | ~90 Gb/sample |
| Turnaround time | ~7 weeks after DNA QC |
| Sample type | gDNA |
++ DNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- DNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
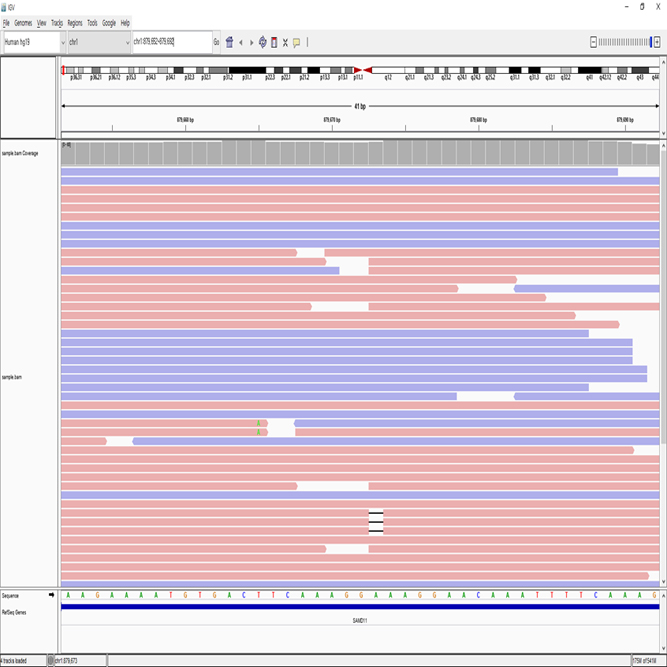
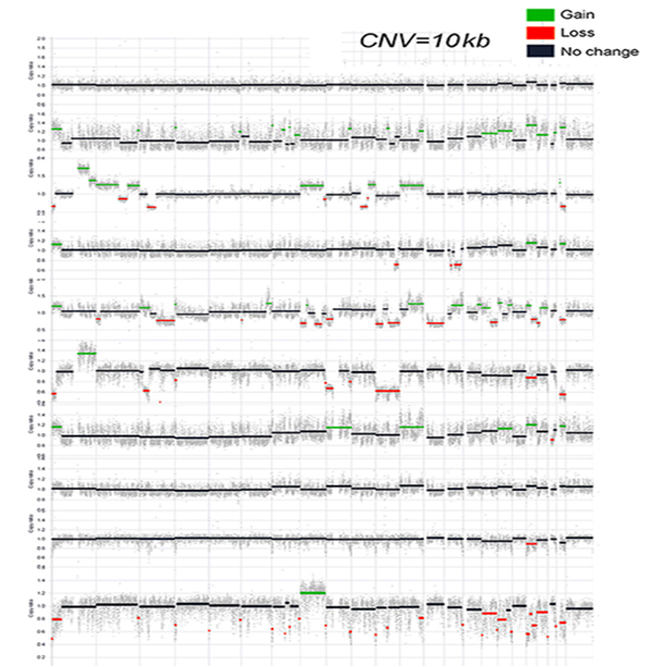
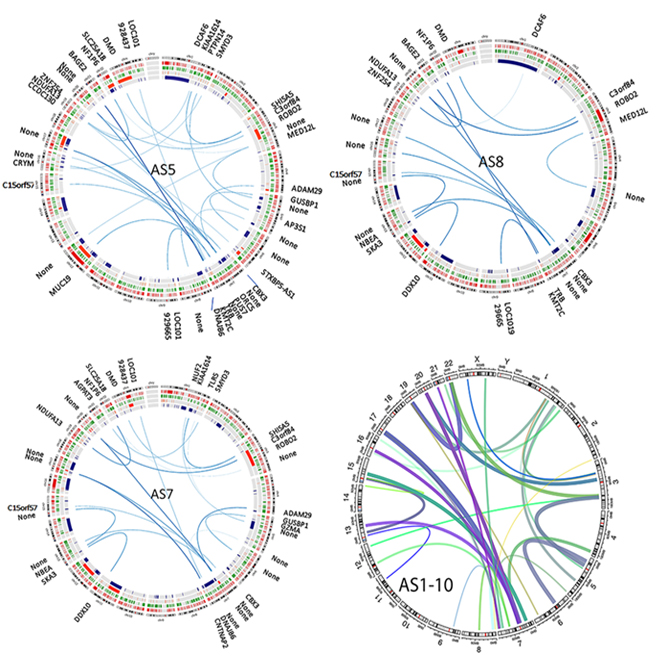
SNP(Single Nucleotide Polymorphism), INDEL (Insertion & Deletion), CNV(Copy Number Variation), Structural Variation 등의 다양한 분석을 지원해 드립니다.
Whole Exome Sequencing은 Exon 영역만을 선택적으로 capture하여 library를 제작하여 엑솜영역의 유전체를 분석하는 방법입니다. 기본 분석으로써 SNP, INDEL, CNV, Structural variation 분석 데이터를 제공하고 이외 다양한 분석을 지원해 드립니다.
Service Info.
| Sample requirement | >2ug gDNA |
|---|---|
| Library method | Agilent SureSelect kit |
| NGS run format | NovaSeq 6000, PE100 |
| Data yield | ~6 Gb (100X depth)/sample |
| Turnaround time | ~6 weeks after DNA QC |
| Sample type | gDNA |
++ DNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- DNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
SNP(Single Nucleotide Polymorphism), INDEL (Insertion & Deletion), CNV(Copy Number Variation), Structural Variation 등의 다양한 분석을 지원해 드립니다.
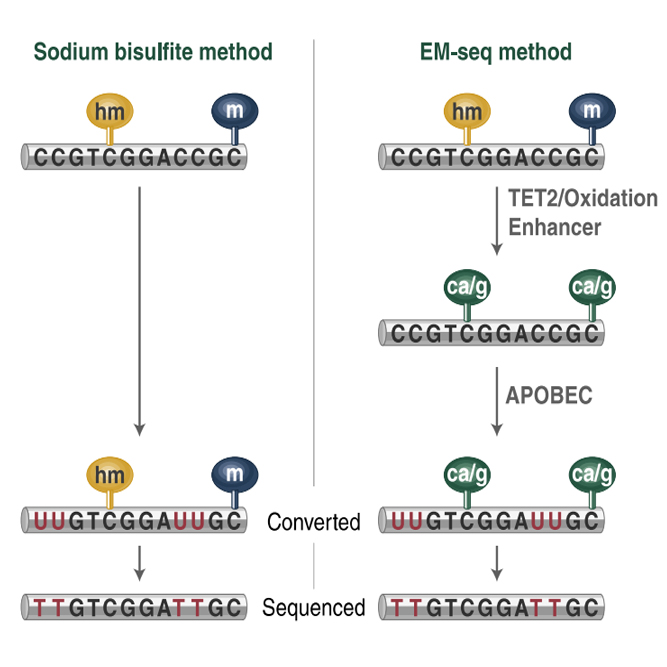
Enzymatic Methyl-Seq(EM-Seq)은 DNA 손상 없이 methylation 분석이 가능한 서비스로 기존의 bisulfite conversion 방식의 Whole Genome Bisulfite Sequencing(WGBS)의 bisulfite처리하기 때문에 DNA가 손상되어 편향된 결과를 얻을 수 있는 단점을 enzyme-based 방법을 사용하여 DNA의 손상을 최소화하여 효과적으로 5-mC와 5-hmC를 검출 할 수 있는 서비스를 제공해드립니다.
Service Info.
| Sample requirement | >2ug gDNA |
|---|---|
| Library method | NEBNextⓇ Enzymatic Mehyl-seq kit |
| NGS run format | Illumina NovaSeq6000, PE150bp |
| Data yield | ~120G/sample |
| Turnaround time | ~6 weeks after DNA QC |
| Sample type | gDNA |
- DNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
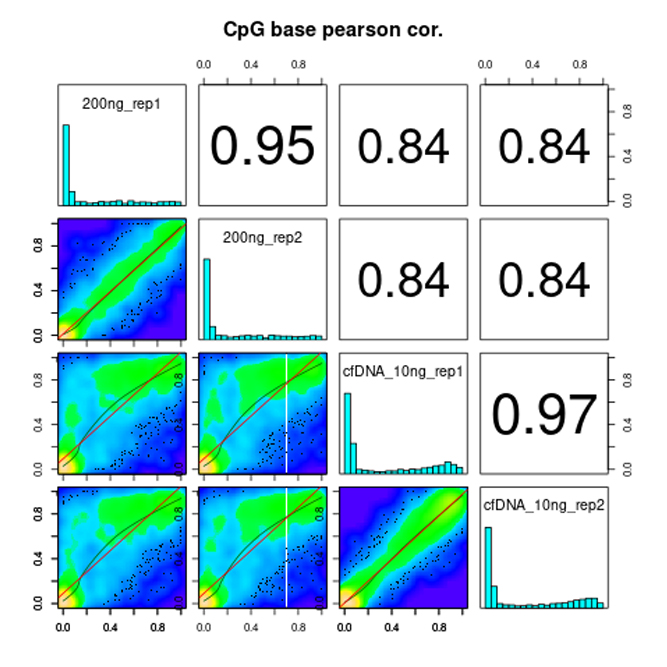
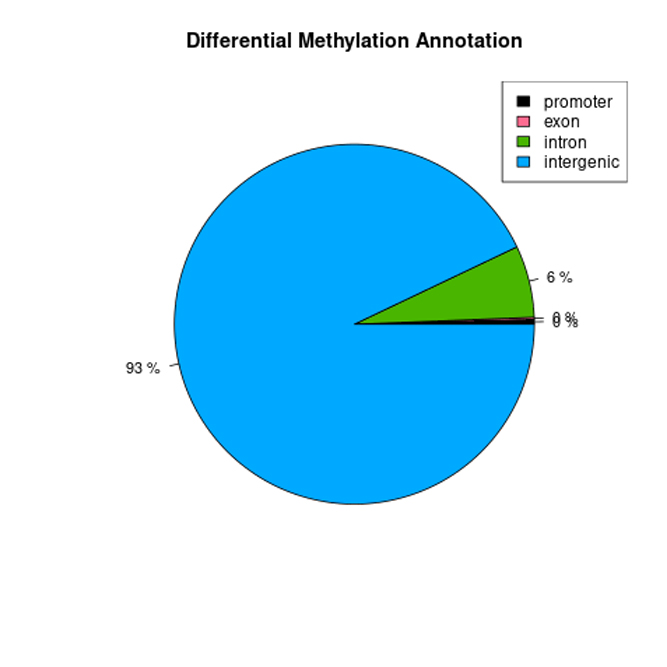
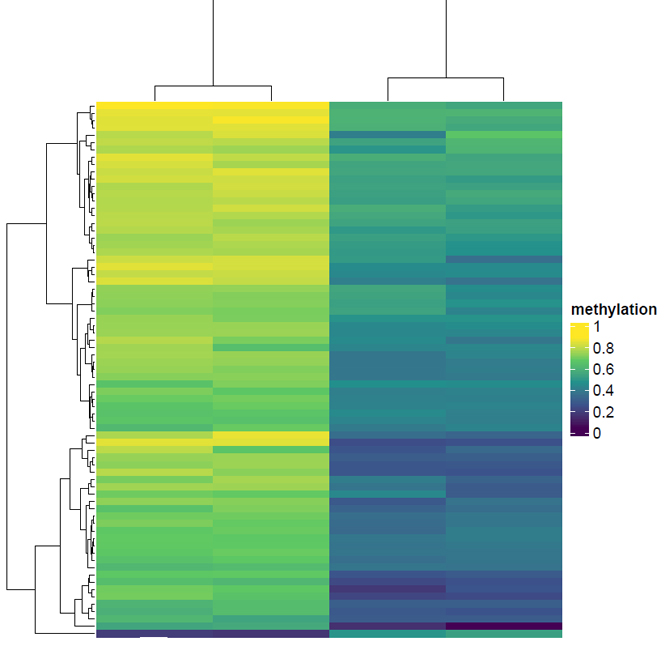
ExPADA Report, Methylation Calling, Differential Methylated Cytosines(DMC) Analysis & Annotation, Differential Methylated Regions(DMR) Analysis & Annotation, Sample Correlation & PCA plot 등 다양한 분석을 지원해 드립니다.
DNA와 조절단백질과의 상호작용을 분석하고 후생유전학 연구를 위해 매우 유용한 ChIP-Seq 서비스를 제공해 드립니다. 오랜 기간의 실험 노하우와 분석기법을 통해 연구자가 필요로 하는 데이터를 제공해 드립니다.
Service Info.
| Sample requirement | >20ul IP-DNA |
|---|---|
| Library method | NEB Ultra DNA Library kit |
| NGS run format | Illumina NovaSeq6000, PE100bp |
| Data yield | ~4G/sample |
| Turnaround time | ~3 weeks after Library QC |
| Sample type | IP-DNA |
- Library Prep.
- Library QC
- NGS
- Data Analysis
- Report
Data Analysis
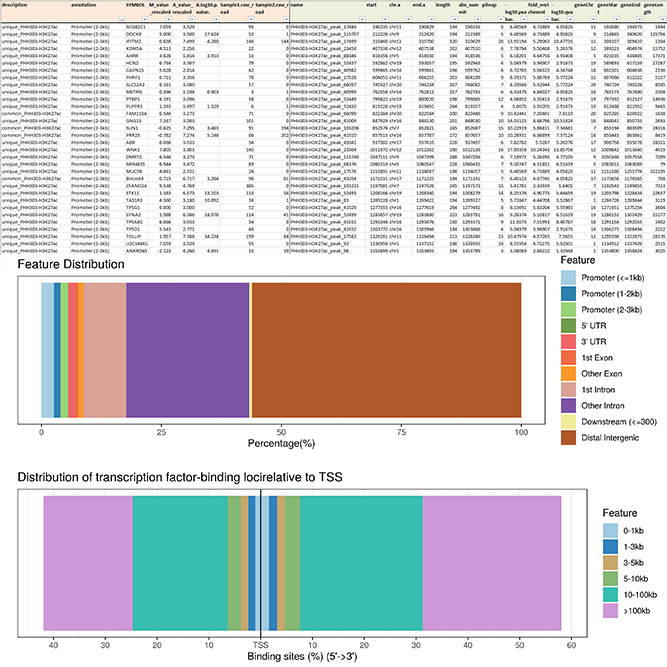
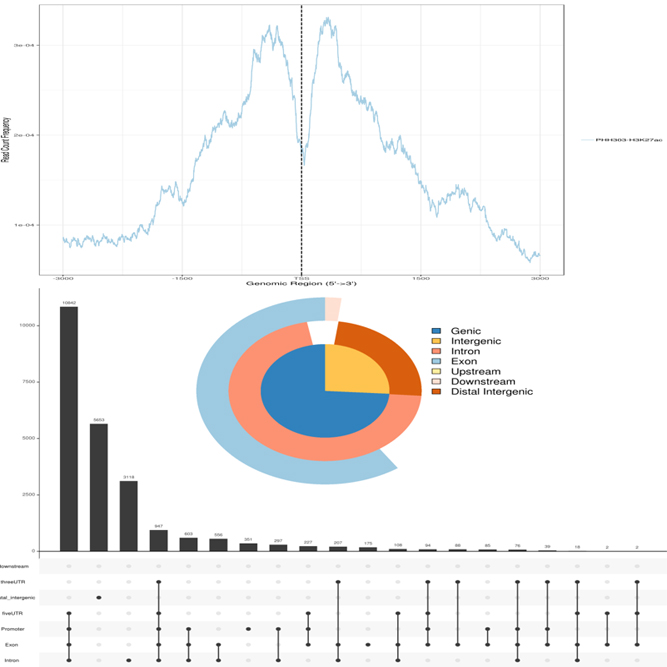
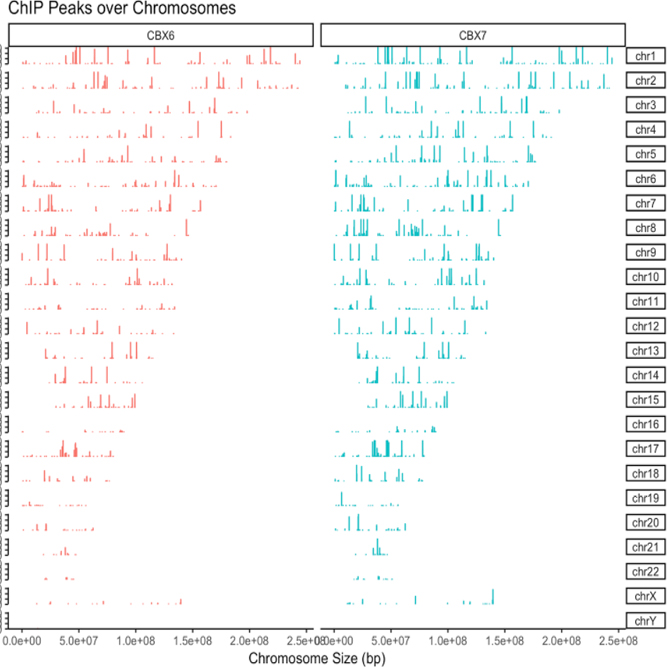
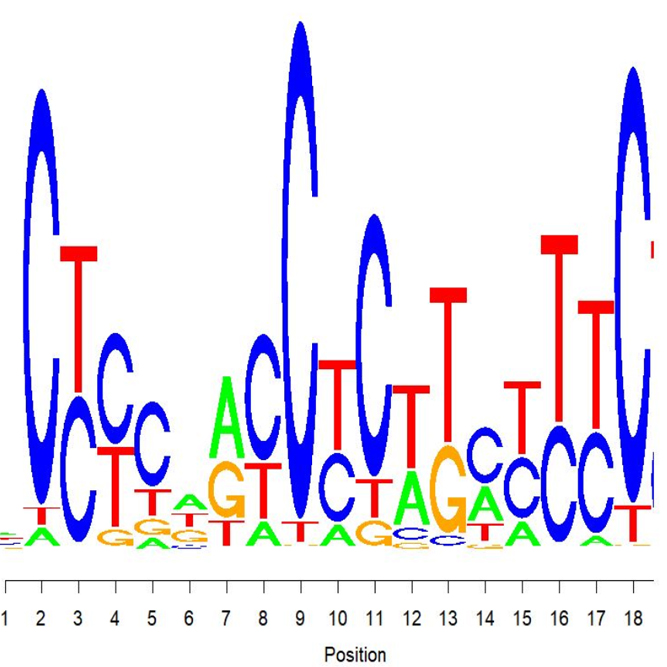
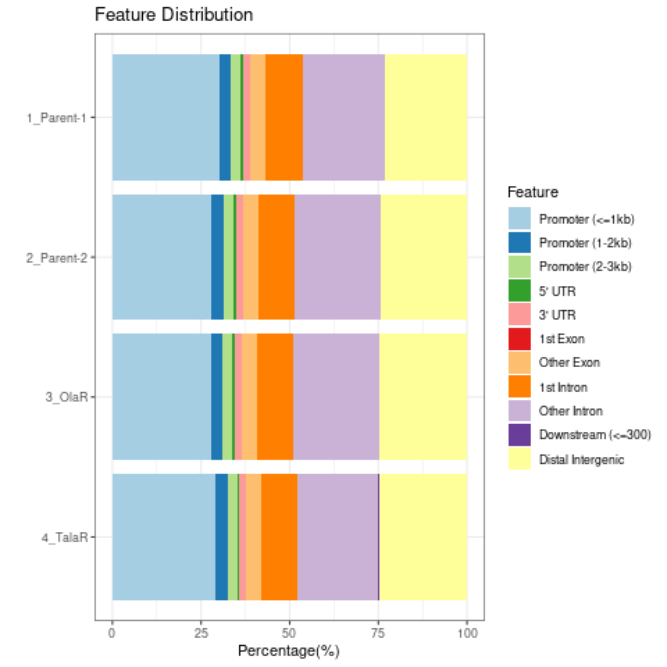
ExDEGA Report, Peak Annotation, Motif Discovery, Tags Comparison, IGV, BedGraph, Data Mining 등의 다양한 분석을 지원해 드립니다.
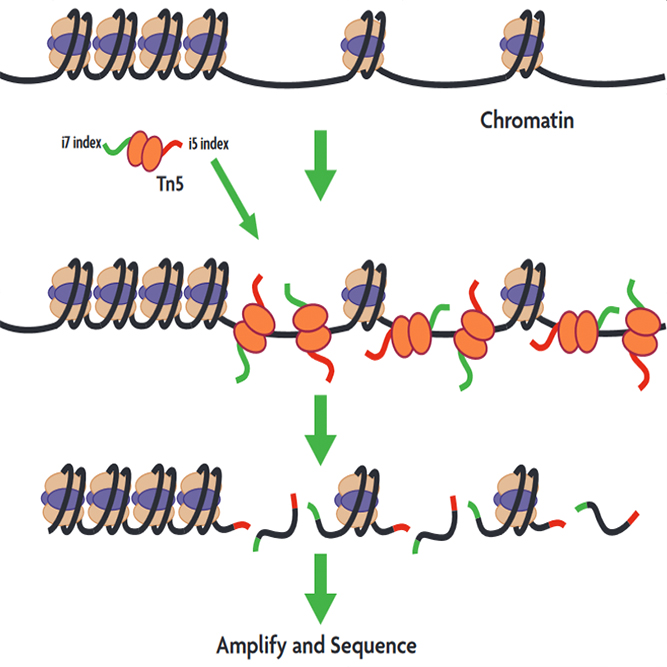
ATAC-seq(Assay for Transposase-Accessible Chromatin using sequencing)은 DNA 발현과정에서 일어나는 chromatin 구조변화인 open chromatin영역만을 자르는 Tn5 transposase을 이용하여 open chromatin영역을 genome-wide로 profiling하는 분석 방법입니다.
Service Info.
| Sample requirement | >~1x106 cells in 1ml media (Cell viability >80%) |
|---|---|
| Library method | ACTIVE MOTIF® ATAC-Seq Kit |
| NGS run format | NovaSeq 6000, PE100 |
| Data yield | ~6G/ sample |
| Turnaround time | ~5 weeks after Cell QC |
| Sample type | Cell |
- Cell QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
ExPADA Report, Peak Annotation, Motif Discovery, Tags Comparison, IGV, BedGraph, Data Mining 등의 다양한 분석을 지원해 드립니다.
토양, 공기, 물, 인체시료 등의 다양한 환경에서 채취되는 시료내에 어떠한 미생물(Bacteria, Fungi 등)들이 얼만큼 존재하는지를 NGS 기술을 적용한 16s rRNA sequencing을 통해 Metagenome / Microbiome 분석을 수행해 드리고 있습니다.
Service Info.
| Sample requirement | >0.1 ng/ul gDNA |
|---|---|
| Library method | 16S rRNA Amplicon Library for illumina |
| NGS run format | Illumina MiSeq, PE300bp(Paired End 300bp) |
| Data yield | ~50,000 reads/sample |
| Turnaround time | ~4 weeks after Library QC |
| Sample type | gDNA |
++ DNA prep.을 원하실 경우 담당자와 상의하여 진행이 가능합니다.
- DNA QC
- Library prep.
- NGS
- Data Analysis
- Report
Data Analysis
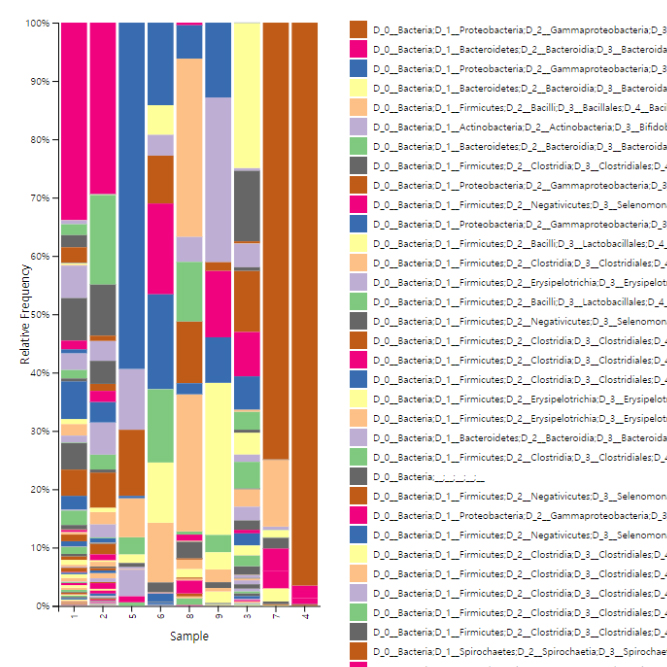
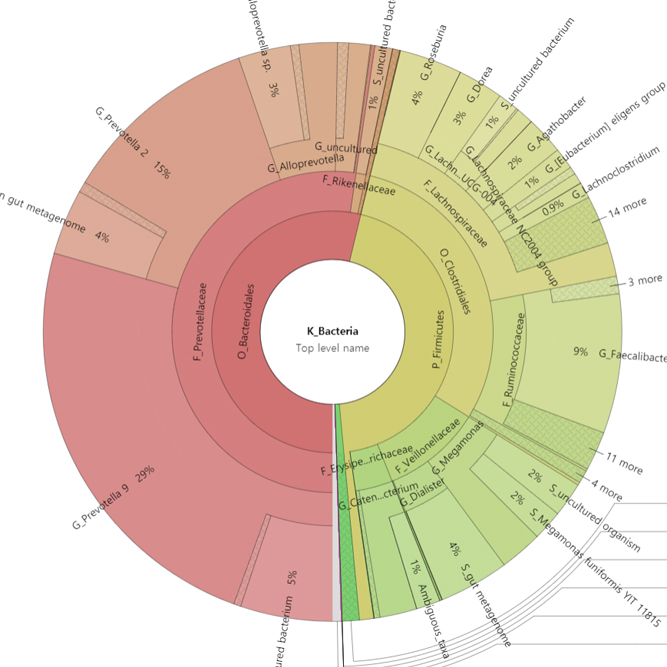
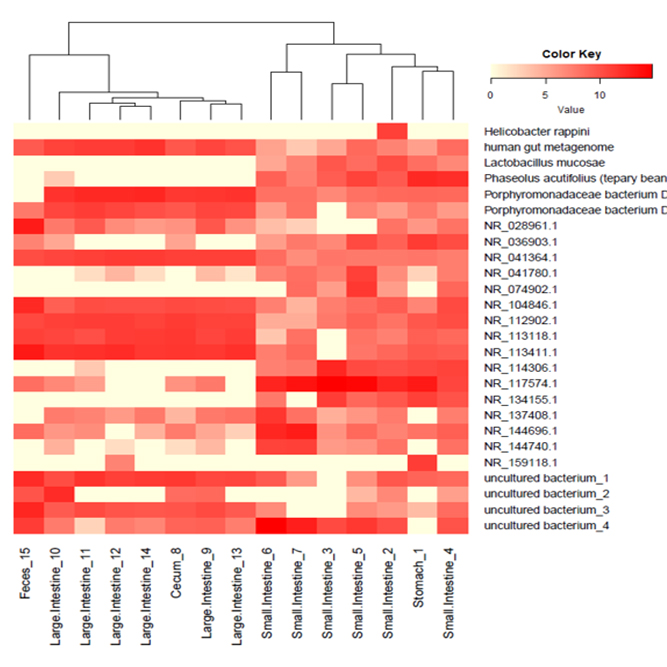
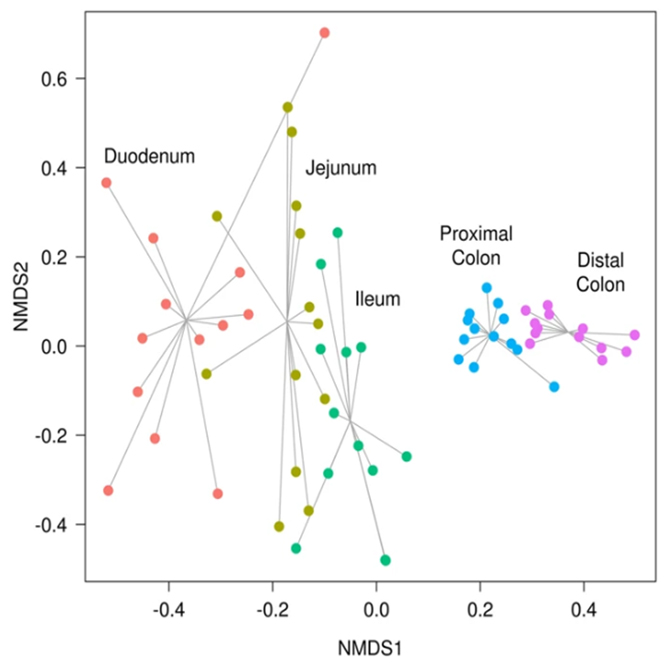
Assembly Result, Feature Table, Taxonomy(Bar & Pie Chart, Profile Data), Diversity(Alpha, Beta), Sample Clustering 등의 다양한 분석을 지원해 드립니다.